一直以来,对生物样本(包括细胞系、类器官、异种移植和同种移植模型)进行验证都是研究中的重要步骤,但验证错误和污染仍是亟待解决的一个问题。本文中,我们探讨了生物样品验证和污染评估的两种传统的(低通量)基于基因组的检测方法:短串联重复序列(STR)和单核苷酸多态性(SNP)分析。另外,本文也强调了深度下一代测序(NGS)作为生物样品验证和污染检测的高通量和多功能方法的价值,NGS与两种传统方法相比,具有更高的灵敏度。
为什么生物样本验证很重要?
在生命科学研究中,细胞系一直发挥着重要作用,但研究表明,细胞系错误和污染时有发生。例如,交叉污染和错误标记都可能造成细胞系的错误识别。国际细胞系验证委员会(ICLAC)有个名单,记录了多达530多个错误识别的细胞系(2021年6月更新),且名单数量还在不断增加。
使用错误识别的细胞系是一个越来越严峻的问题,一项研究发现,超过32,500篇论文涉及上述ICLAC列出的错误识别的细胞系。
污染也是一个日益严重的问题。污染的来源多种多样,包括与其他细胞系和微生物(例如支原体和/或病毒)的交叉污染。总的来说,估计有:
- 大约 15-20% 的细胞系与其他细胞系发生了交叉污染或错误识别
- 5-30% 的细胞系可能被支原体污染
- 超过 25% 可能被至少一种病毒污染
无论问题的根源是什么,使用错误识别和/或受污染的细胞系得到的研究结果都可能是不可信的,并由此导致结论错误,时间、精力和金钱的浪费,以及声誉受损。这就是为什么基金会和越来越多的出版方都要求研究人员提供他们的细胞系和其他生物样本的验证。
鉴于使用真实和干净的生物样本的重要性,研究人员应考虑对他们的样本进行验证:
- 当新获得或新开发生物样本时应进行验证
- 验证应作为标准的质量控制措施
- 当有疑问时应进行验证
生物样品验证的常规方法及其局限性
短串联重复(STR)分析
STR分析使用的引物可以识别长度为2-6个碱基对的重复DNA片段。由于目标片段在给定人群中有所不同,由此就可生成DNA“指纹”。该技术对于验证细胞系和追踪源自患者的异种移植或细胞系人源肿瘤样本具有重大价值。
美国组织培养物保藏中心(ATCC)曾发布了详细的指南,介绍如何以标准化STR分析进行人源细胞系鉴定。据报道,STR检测的灵敏度为5-10%,灵敏度受到被测细胞系的性质和数据质量的影响。
STR作为一种常用技术,是目前生物样本验证的“金标准”,但其使用也存在一些众所周知的限制:
- 低通量:这使得大批量样品的验证变得非常繁琐
- 单功能:需要其他检测来评估其他参数(例如,检查生物样本中的细胞系和肌原体污染需要使用不同的检测)
- 劳动密集:验证大批量样品成本高昂
- 检测污染时灵敏度变动:检测污染的能力不一致
此外,已知具有错配修复(MMR)基因突变的生物样品会表现出微卫星不稳定性和超突变表型。这会导致遗传漂变和/或污染细胞的生长以及STR错误分类。在遗传关系密切的情况下,STR测定的准确性也可能较低,例如验证来自同一人类供体的不同肿瘤细胞系或源自缺乏独特遗传标记的特定近交动物品系的鼠源细胞系。
基于多重PCR/qPCR的SNP分析
与依赖核苷酸重复的STR分析相反,SNP分析评估单个DNA核苷酸水平的变异。SNP是指特定位点的单碱基对突变或单核苷酸多态性,通常由两个等位基因组成。尽管这些都是自然发生的变体,但已发现一些SNP与许多人类疾病的病因有关,并且在药物基因组学中具有重要意义。
近年来,SNP基因分型因其更高的准确性、灵敏度和更低的成本而越来越多地用于细胞系和生物样品的验证。SNP可以克服与STR分析相关的一些限制,例如,可用于验证物种特异性肿瘤模型,甚至是MMR缺失的人源肿瘤细胞系。
然而,和STR一样,传统的SNP检测采用多重PCR/qPCR方法,验证大量样本仍然通量较低且繁琐。此外,随着多重PCR/qPCR的增加,覆盖SNP数量(覆盖范围)变得更具有挑战。尽管报道称这些SNP检测的一般灵敏度为3-5%(类似于STR),但研究表明灵敏度取决于样本,且通常低于声称值。
虽然STR和传统SNP分析还继续应用于生物样品验证,但测序技术的进步已经让现在的研究人员能够以非常高的灵敏度进行高通量分析。这种方法在肿瘤学领域很有意义,该领域一直处于可重复性危机的焦点,并且大型生物样本库(例如类器官和患者来源的异种移植[PDX])的使用也在快速增长。使用传统的基于STR和SNP的身份验证方法有一定的劣势。
基于NGS的SNP分析
NGS技术克服了与传统生物样本验证方法相关的许多常见问题,带来了基因组学的变革。
条码深度NGS(BarcodedeepNGS)使用多重PCR扩增目标DNA区域(也就是所谓的“条码”)以进行高深度的测序。当与强大的统计分析相结合时,可以识别出独特的SNP指纹,从而将不同样品区分开。NGS不仅可以检测大量SNP,其技术进展也提高了成本效率和技术准确性。
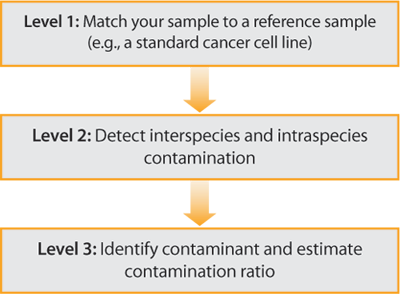
如下图所示,基于NGS的方法,冠科生物可进行三个级别的生物样本验证:
- 第1级:将样本与参考物(例如癌细胞系)进行匹配。我们采用NGS的方法为人类样本提供了200个SNP位点的高深度(3000倍)测序,并且在识别样本或识别受污染样本的主要成分方面显示出100%的准确度。相对传统的STR/SNP有显着进步。
- 第2级:检测生物样品中的污染。我们的方法在使用异质性比率时始终达到2%的灵敏度,如果污染物在具有SNP指纹的参考样本库中,则灵敏度可以达到≤1%。
- 第3级:识别受污染样品中的污染成分。细胞系的交叉污染在生物库中很常见,并且由于细胞系的生长速度不同,受污染培养物的组成会随着时间的推移而发生变化。我们的SNP指纹库由1000多个癌细胞系组成,能可靠地识别污染细胞系。此外,我们的方法还提供了污染率的准确估计。
最近的一项研究证实了基于NGS的验证与传统的基于STR和SNP的方法相比的优势。
对于第1级验证,该研究表明,在高深度(3000倍覆盖率)下对200个SNP位点进行测序,对样品或主要成分识别具有100%的准确度。并且一次运行可以分析数百个样品,相比于低通量的传统STR/SNP检测是重大进步。
对于第2级验证,灵敏度始终达到2%,在某些情况下低于或等于1%,这接近此类检测的理论检测极限,与传统STR/SNP方法相比,变异更小了。
第3级验证涉及污染物的识别,是目前其他方法无法实现的。基于NGS的SNP分析和先进的统计方法使这项功能成为可能。此外,NGS测序还可以检测人-鼠种间污染。由于PDX将人类肿瘤细胞转植给小鼠,使得肿瘤包含小鼠的基质细胞,给STR分析带来困扰。NGS测序时可以使用具有相同旁侧序列的不同DNA片段代替SNP,就能准确检测种间污染成分。
NGS的另一个优势在于检测支原体污染,这是STR或传统SNP检测无法进行的。使用通用引物或物种特异性引物,可以相对轻松地对所有或个别支原体进行靶向测序。
下表比较了两种传统验证技术与基于NGS的验证技术。
| 检测方法比较 | 基于 NGS的SNP | STR | SNP |
|---|---|---|---|
| 技术 | 条码深度 NGS | 多重 PCR和毛细管电泳 | 多重 PCR/qPCR |
| 读数类型 | 数字信号(干净、接近零的定量误差) | 模拟信号(嘈杂、量化误差高) | 模拟信号(嘈杂、量化误差高) |
| 人源样本验证 | 可以 | 可以 | 可以 |
| 鼠源样本验证 | 可以 | 有限度 | 不可以 |
| MMR缺失细胞系验证 | 可以 | 不可以 | 可以 |
| 污染检测灵敏度 | 高(1%) | 中低(5-20%) | 中低(3-20%) |
| 准确性 | 高 | 中低 | 中低 |
| 通量 | 高 | 低 | 低/td> |
| 污染物验证 | 可以 | 不可以 | 不可以 |
| 污染物比例定量 | 可以 | 不可以 | 不可以 |
| 适用于大型生物库 | 可以 | 不可以 | 不可以 |
| 种间污染物检测 | 可以 | 有限度 | 有限度 |
| 种内污染物检测 | 可以 | 有限度 | 有限度 |
| 无参考检测污染物 | 可以 | 不可以 | 不可以 |
| 3个以上细胞系之间的污染比例检测 | 可以(灵敏度1%) | 不可以 | 不可以 |
总体而言,基于NGS的SNP分析是目前唯一可以实现3级验证的商业化分析方法,并且在第1级和第2级验证中优于传统的STR/SNP分析。与传统的STR和SNP基因分型相比,NGS分析更全面,通量更高。
总结
随着基金会、期刊和科学界都要求研究人员证明其生物样本的真实性,对小鼠和人类生物样本(包括细胞系、类器官、异种移植和同种移植模型)的验证变得越来越重要。
虽然基于STR和SNP的传统验证方法曾经非常有效,也将继续在生物样品验证中发挥作用,但显然也需要具有更高灵敏度和更高通量的新技术来克服一些限制,满足生物样本库和先进研究模型的需求。
基于NGS的SNP分析是一种用于细胞系和模型验证的高通量和相对低成本的方法。非常适合建立和维护生物库以及对种间污染和微生物群污染进行高级分析。
冠科生物独特的基于NGS的SNP基因分型组合包含600多个SNP和染色体片段,可准确刻画小鼠和人类的各种样本,包括细胞系、类器官、异种移植模型和患者组织。我们还为最常用的肿瘤研究模型生成了独特的DNA指纹,涵盖1,200多种人类癌症和30种同源细胞系。
如果您有兴趣了解更多关于如何使用基于NGS的分析来满足您生物样品验证的需求,请观看我们最近的网络研讨会 Advancing Authentication: Next Generation Technology for Cell Line and Biosample Verification,或请立即联系我们。